Красивая визуализация геномных аннотаций с DNA Features Viewer
МЕНЮ
Искусственный интеллект
Поиск
Регистрация на сайте
Помощь проекту
ТЕМЫ
Новости ИИ
Голосовой помощник
Городские сумасшедшие
ИИ в медицине
ИИ проекты
Искусственные нейросети
Слежка за людьми
Угроза ИИ
Компьютерные науки
Машинное обуч. (Ошибки)
Машинное обучение
Машинный перевод
Нейронные сети начинающим
Реализация ИИ
Реализация нейросетей
Создание беспилотных авто
Трезво про ИИ
Философия ИИ
Генетические алгоритмы
Капсульные нейросети
Основы нейронных сетей
Распознавание лиц
Распознавание образов
Распознавание речи
Техническое зрение
Чат-боты
Авторизация
2020-08-07 09:58
В Python существует множество разных биоинформатических пакетов, необходимых для анализа данных. Но данные нужно не только хорошо и правильно анализировать, но и красиво визуализировать, чтобы другие могли их быстро и легко понять, а также получить эстетическое удовольствие от созерцания.
Библиотека DNA Features Viewer позволяет визуализировать геномные аннотации, например, показать проаннотированный элемент, его тип, начало и конец, название, виды, у которых встречается, и другие характеристики. В том числе, эта библиотека позволяет пользователю менять цвет, шрифты и представлять данные так, как хочется.
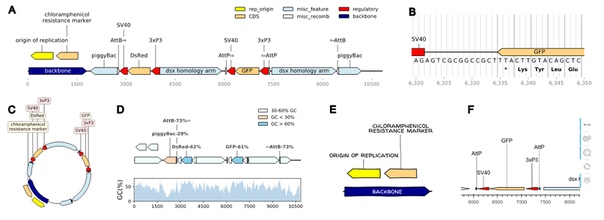
На самом деле, визуализация данных – очень важная часть в биоинформатике, и, конечно, есть множество способов, которые позволяют это делать. Как примеры таких программ (и в копилку для вас), можно указать SnapGene Viewer или Benchling, которые дают возможность выделять цветом, что-то показывать, а что-то скрывать из визуализации аннотации, но не могут быть автоматизированы и количество опций в них всё же ограничено. Существует ещё gff2ps, а также некоторые библиотеки для Python, например, DnaPlotLib и Biopython, в которых всё равно приходится над каждой аннотацией работать отдельно, а также в них, к сожалению, нет автоматических правок ситуаций, когда фрагменты рисунка или подписи перекрываются.
Со всем этим справляется библиотека для Python, о которой мы рассказываем – DNA Features Viewer. Она использует аннотации из GFF файлов, Biopython SeqRecords, либо Genbank, которые переводит в так называемые graphic records, определяющие визуальные характеристики каждой аннотации. Именно с ними уже и можно работать и приводить визуализацию к желаемому виду. Класс, который производит перевод аннотации, позволяет выбирать что показывать, а что – нет, определять цвет и другие параметры объектов на рисунке. Сохранять полученные схемы можно в форматах PNG, SVG, PDF и ещё некоторых других.
Ссылка на GitHub – https://github.com/Edinburgh-Genome-Foundry/DnaFeaturesViewer. Кстати, здесь сразу есть примеры кода и картинок, которые с его помощью создаются. Очень полезно!
Ссылка на документацию – https://edinburgh-genome-foundry.github.io/DnaFeaturesViewer/
Ссылка на статью – https://academic.oup.com/bioinformatics/article-abstract/doi/10.1093/bioinformatics/btaa213/5868559.
Источник: m.vk.com