Как понять, что ваши гены отбирают?
МЕНЮ
Искусственный интеллект
Поиск
Регистрация на сайте
Помощь проекту
ТЕМЫ
Новости ИИ
Голосовой помощник
Городские сумасшедшие
ИИ в медицине
ИИ проекты
Искусственные нейросети
Слежка за людьми
Угроза ИИ
Компьютерные науки
Машинное обуч. (Ошибки)
Машинное обучение
Машинный перевод
Нейронные сети начинающим
Реализация ИИ
Реализация нейросетей
Создание беспилотных авто
Трезво про ИИ
Философия ИИ
Генетические алгоритмы
Капсульные нейросети
Основы нейронных сетей
Распознавание лиц
Распознавание образов
Распознавание речи
Техническое зрение
Чат-боты
Авторизация
2020-07-05 12:05
Статья Дмитрия Бибы для «Биомолекулы» расскажет о том, как и зачем искать следы естественного отбора в геномах живых организмов, каким бывает естественный отбор и о чем говорит наличие или отсутствие его действия на гены. Также статья коснется вопроса о пользе этой деятельности для человечества.
Эволюция — это то, что вдыхает смысл в биологию («Нет, — сказал я себе, — я не буду начинать статью с цитаты Добржанского...»). А главный двигатель эволюции, как придумал Дарвин, это естественный отбор. Поэтому, если хочется что-то понять про эволюцию, в большинстве случаев это значит, что надо понять что-то про естественный отбор.
Самые ранние свидетельства работы естественного отбора люди наблюдали на уровне фенотипа — вы наверняка помните бабочек в Британии, становившихся в череде поколений все более темными на территории загрязненного леса [1]. А если все-таки не помните, вкратце, история такая: в середине XIX века в Англии из-за промышленного загрязнения воздуха лишайники на деревьях стали умирать, и темные бабочки на фоне стволов стали менее заметными для птиц, поэтому начали побеждать светлых бабочек в «борьбе за существование». Наблюдая отбор в реальном времени, можно понимать, какие признаки организмов важны для их выживания и размножения, — это те признаки, которые распространяются в популяции.
Но посмотреть на отбор в реальном времени удается далеко не всегда. Хорошо наблюдать за бабочками — они живут сильно меньше человека. А попробуйте понаблюдать за оленями! За распространением признака в их популяции можно наблюдать всю жизнь. Это если еще повезет угадать, на какой признак надо смотреть — ведь процесс этот настолько медленный, что с первого взгляда и не поймешь, идет ли он вообще.
В наше время, однако, жизнь эволюционного биолога стала намного проще. Теперь можно смотреть на гены, определяющие признаки. А в генах заключено очень много информации! Они несут на себе отпечаток действовавшего или действующего на них отбора. Если мы понимаем, на какие участки генома действует отбор, мы можем сделать вывод о том, какие гены, и связанные с ними признаки, важны для выживания и размножения их носителей.
Зачем это нужно? Ну, прежде всего, это интересно. Например, интересно ведь узнать, какие гены делают человека человеком (то есть экспрессия или наличие каких генов отличает человека, скажем, от шимпанзе). Предположительно, те, на которые у человека действовал особый тип отбора — положительный (что это значит, я очень скоро объясню). Но есть и более насущные приложения знаний об отборе на уровне генов. Если, например, я разрабатываю антибиотик против какого-то вида бактерий, на какие бактериальные белки я его хочу натравить? На которые действует другой тип отбора — отрицательный (опять же, объяснение чуть ниже). Или, может, мне интересно, за счет чего крысы, исправно умиравшие от применения какого-нибудь яда, вдруг перестали это делать? Опять же ищем, на какие гены действовал положительный отбор у крыс, которых мы травили. Или рак. Как это поможет в лечении рака? Пожалуйста! Раковая опухоль — фактически, популяция одноклеточных организмов. «Успешная» опухоль состоит из клеток-носителей генов, на которые действовал положительный отбор. Если мы понимаем, на какие гены действует положительный отбор в раковых опухолях, мы можем обращать внимание на мутации в этих генах и предсказывать риск раковых заболеваний у пациентов до их появления и принимать превентивные меры.
Типы отбора
Существует не одна классификация типов естественного отбора (например, [2]). Я буду пользоваться делением на положительный, отрицательный и балансирующий.
- Отрицательный отбор — самый распространенный в природе. Это когда у гена все хорошо, и (почти) никакие изменения ему не полезны. Если в гене происходит мутация, она, скорее всего, портит его, и зверь с этой мутацией оставляет меньше потомков, чем зверь без мутации (окей, давайте сделаем паузу здесь: каждый раз, когда я говорю «зверь», я имею в виду «особь» или «индивид», и это никак не связано с систематикой). Поэтому мутация исчезает из популяции.
- Положительный отбор — самый интересный. Это когда гену есть к чему стремиться. Если в гене происходит мутация, она может быть полезной, и зверь с этой мутацией оставляет больше потомков, чем зверь без мутации. Поэтому мутация распространяется в популяции и фиксируется (закрепляется, то есть все члены популяции становятся ее носителями). Между прочим, положительный отбор, приведя к фиксации мутации, становится отрицательным: теперь изменения в соответствующей позиции будут вредны и станут отсеиваться отбором. Почему положительный отбор самый интересный? Потому что именно он стоит за адаптациями — полезными признаками, приобретаемыми организмом. А биологи очень любят рассуждать о том, почему те или иные признаки полезны и полезны ли они вообще.
- Балансирующий отбор — самый... неоднозначный. Это когда ген не вполне уверен, чего он хочет — ни одна мутация не может считаться однозначно полезной или вредной. Например, мутация считается полезной только пока она редкая (звери с мутацией оставляют больше потомков, чем звери без мутации, только пока зверей с этой мутацией мало). Поскольку в момент своего появления мутация еще редка, она начнет распространяться в популяции. Но с течением времени она будет становиться все более частой, и значит, все менее полезной. Поэтому при превышении некоторого порога частота мутации начнет падать. Получается, что мутация не фиксируется, но и не исчезает. Неоднозначность балансирующего отбора состоит в том, что существуют серьезные сомнения в его распространенности в природе. Впрочем, возможно, что его не встречают, потому что плохо умеют искать (а искать балансирующий отбор действительно непросто).
Все типы отбора интересно идентифицировать в геноме для разных целей.
Отрицательный отбор
Я уже сказал, что он встречается в геноме чаще всего. Это, в принципе, интуитивно понятно. Ведь не просто так у нас 20 тысяч генов, они нам для чего-то нужны. Кодируют какие-то белки. Белки выполняют какие-то функции, и выполняют в основном хорошо. Поэтому мутации, возникающие в кодирующих их генах, скорее всего, приводят к ухудшению выполнения этими белками своих функций. И все-таки, как в этом убедиться? Может, большинство генов нам и нужны такими, какие они есть, но могу ли я быть уверен, что какой-то конкретный ген мне зачем-то нужен именно таким, и на него действует отрицательный отбор?
Да, могу. И, на самом деле, история об этом очень тесно связана с историей о положительном отборе.
Статистика dN/dS
Стратегия будет такой.
- Поймем, что происходит, когда отбора нет.
- Посмотрим туда, где ожидаем увидеть или не увидеть отбор.
- Если там происходит то же самое, что и в случае, когда отбора нет... значит, отбора нет. Поразительно.
- Если там происходит что-то другое, отбор есть. Какой? Смотря что происходит.
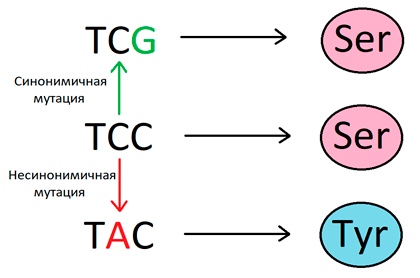
На что бы нам посмотреть, чтобы понять, что происходит, когда отбора нет? Самое безопасное, что мы можем сделать — посмотреть на синонимичные мутации (такие, которые не меняют аминокислотный состав белка) (рис. 1). С некоторыми оговорками (например, с такими — [3]), отбор в них происходить никак не должен — зверь с синонимичной мутацией никак не отличим от зверя без нее, пока мы не заглянем в его геном.
Процесс, на который мы будем смотреть и который поможет отличить наличие отбора от его отсутствия — это фиксация мутаций. Проследить его можно, сравнивая один и тот же ген у двух видов (рис. 2). Скажем, у человека и шимпанзе. Если мы видим, что в какой-то позиции у всех людей написано одно, а у всех шимпанзе — другое, мы говорим, что произошла замена (хотя, честно говоря, чтобы понять, у кого же из них она произошла, понадобится третий вид — например, орангутан). Она может быть синонимичной (не приводить к замене аминокислоты) или несинонимичной (приводить).
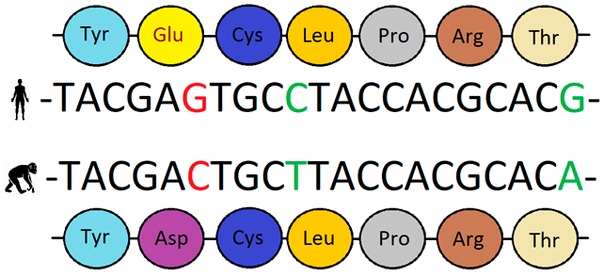
Посмотрим сначала на синонимичные замены. Отбор на них не действует (помните, однако, про оговорки!), поэтому они происходят только в силу случайных причин (этот процесс называется генетическим дрейфом).
Теперь посмотрим на несинонимичные замены. Это как раз третий шаг нашего плана — на несинонимичные позиции отбор мог действовать или не действовать. Если несинонимичные замены происходят с той же частотой, что и синонимичные, это значит, что вероятность фиксации мутации никак не зависит от того, меняет она аминокислоту в белке или нет. То есть мутации в этом гене никак не влияют на приспособленность организма. Отбора нет. Такое называют нейтральной эволюцией, и она встречается в псевдогенах — сломанных генах (об этом можно почитать статью на «Биомолекуле»: «Как нонсенс-мутации ген обижали, и что потом было» [4]). Но если несинонимичные замены происходят реже, чем синонимичные, то, видимо, вероятность закрепиться для несинонимичной мутации ниже, чем для синонимичной. Другими словами, среди несинонимичных мутаций встречаются вредные. Это значит, действует отрицательный отбор, отсеивающий эти мутации. Если же, наоборот, несинонимичные замены происходят «подозрительно часто» (чаще, чем синонимичные), это значит, что среди несинонимичных мутаций встречаются полезные с повышенной вероятностью закрепления. Значит, действует положительный отбор (посмотрите также рис. 3).
Если обозначить частоту несинонимичных замен как dN, а частоту синонимичных — dS, все эти рассуждения можно выразить кратко вот так:
dN = dS, или dN/dS = 1 ? нет отбора
dN < dS, или dN/dS < 1 ? отрицательный отбор
dN > dS, или dN/dS > 1 ? положительный отбор
Эта оценка — dN/dS — была проделана для большинства генов множества видов. Оказалось, что, действительно, для большинства генов она много меньше единицы, что свидетельствует о преобладании отрицательного отбора. Surprise-surprise: гены нам для чего-то нужны такими, какие они есть.
Положительный отбор
Казалось бы, говорить уже не о чем. Когда метрика dN/dS > 1, тогда и действует положительный отбор, дело закрыто. Но нет. Дело в том, что под положительным отбором обычно находится лишь небольшой участок гена, в то время как оставшаяся его часть остается под отрицательным отбором. Действительно, изменения условий редко когда требуют полной перестройки белка — скорее, небольших изменений. Например, раньше белку нужно было связывать одно вещество, а теперь — другое. Изменить нужно только аминокислоты в центре связывания. Статистика dN/dS говорит нам о том, какой отбор «в среднем» действует на ген. И ответ чертовски предсказуем — в среднем действует всегда отрицательный! И действительно, вы найдете довольно мало генов, для которых dN/dS > 1 — вездесущий отрицательный отбор заглушает редкие сигналы положительного.
Один из примеров гена, находившегося у наших предков под таким сильным отбором, что это заметно по dN/dS — это вариант гена лактазы, фермента, расщепляющего лактозу в молоке, сохраняющийся у взрослых [5]. Другие примеры — гены, экспрессирующиеся при сперматогенезе, помогающие будущим половым клеткам избежать апоптоза [6], и гены иммунного ответа [7], находящиеся под постоянным положительным отбором из-за гонки вооружений с патогенами.
Значит, нужно каким-то разумным образом ослабить порог dN/dS, при превышении которого мы будем считать ген положительно отбираемым.
Критерий Макдональда—Крейтмана
Разумный способ был найден [8]. Стратегия остается такой же: понимаем, как происходит процесс, когда отбора нет — смотрим, как он происходит в интересующем нас месте — если так же, то отбора нет, если по-другому — есть.
Процесс — это все еще закрепление мутаций. Для этого все еще нужно сравнивать один и тот же ген у двух (а лучше трех) видов и смотреть на замены. Но теперь нулевая гипотеза будет другой: при отсутствии отбора синонимичные и несинонимичные замены закрепляются не с одинаковой скоростью, а с какими-то своими, пока неизвестными нам скоростями. Весь трюк состоит в том, чтобы эти скорости оценить.
Чтобы это сделать, посмотрим на интересующий нас ген у разных зверей внутри одного вида. Мы, скорее всего, увидим полиморфизмы — места в гене, в которых у части особей в популяции написано одна буква, а у части — другая. Некоторые из этих полиморфизмов будут синонимичными, другие — несинонимичными (рис. 4).

Понятно, что все синонимичные полиморфизмы нейтральны, ведь на них отбор не действует (оговорки, оговорки; вот, например, такие еще бывают — [9]). Но что можно сказать про несинонимичные полиморфизмы? Мутации в них могут быть полезными, вредными или нейтральными.
Теперь сделаем ход конем. Скажем так: нет в полиморфизмах полезных мутаций (то есть, поддерживаемых положительным отбором). Основания для этого имеются — полезные мутации закрепляются быстрее нейтральных и уж точно быстрее вредных (которые в большинстве своем и вовсе не закрепятся). Поскольку, смотря на полиморфизмы, мы видим лишь моментальный срез во времени, скорее всего, процесс закрепления полезной мутации «в самом разгаре» засечь не удастся. Значит, все несинонимичные полиморфизмы либо нейтральные, либо слабовредные, не успевшие отсеяться отбором.
Теперь возьмем случай, когда отбора нет. То есть все несинонимичные мутации в популяции нейтральны. Интуитивно понятно, что чем их больше, тем больше их и закрепится. То же справедливо и для синонимичных мутаций. Пропорции, в которых мы видим синонимичные и несинонимичные полиморфизмы, — это те же пропорции, в которых они будут становиться заменами. Если синонимичных полиморфизмов в два раза больше, чем несинонимичных, то и синонимичных замен (в отсутствие отбора!) будет в два раза больше, чем несинонимичных. Более общо: при отсутствии отбора синонимичные замены будут происходить во столько раз чаще (реже) несинонимичных, во сколько синонимичных полиморфизмов больше (меньше), чем несинонимичных.
При положительном отборе несинонимичные замены будут случаться чаще, чем «должны», исходя из соотношений полиморфизмов: помимо нейтральных мутаций, которые мы видим, случаются и полезные, которых мы не видим, потому что взяли моментальный срез во времени, и все полезные мутации либо уже закрепились, либо еще не произошли.
При отрицательном отборе некоторые (несинонимичные) мутации, которые мы видим в полиморфизмах, — слабовредные и никогда не закрепятся. Значит, несинонимичные замены будут происходить реже, чем мы этого ожидаем, исходя из количества несинониминых полиморфизмов.
Отношение несинонимичных полиморфизмов к синонимичным обозначается как pN/pS, и соответственно, критерий Макдональда—Крейтмана состоит в следующем:
dN/dS = pN/pS ? отбора нет.
dN/dS < pN/pS ? отрицательный отбор
dN/dS > pN/pS ? положительный отбор
Критерий Макдональда—Крейтмана позволяет обнаружить положительный отбор там, где статистика dN/dS его не видит. С его помощью было обнаружено множество функциональных генов, подвергавшихся положительному отбору у человека [7]. Например, ген ASPM, регулирующий размер мозга при развитии. Интуитивно кажется, что размер мозга — что-то, что должно отличать нас от других приматов, верно? Однако dN/dS этого гена у человека (при сравнении с шимпанзе и орангутаном) получается ~1, что предполагает отсутствие отбора. Не так просто обмануть критерий Макдональда—Крейтмана: pN/pS этого гена в человеческой популяции равен 0,63, то есть, меньше, чем dN/dS, — а это значит, что можно заявлять о положительном отборе на ASPM у человека [11].
Недавний положительный отбор. Да-да, это совсем другое дело
До этого мы искали положительный отбор, действовавший на протяжении всей истории вида. Совсем другое дело — поиск отбора, действовавшего на популяцию недавно. Это относится к примеру про крыс — за счет мутаций в каком гене они перестали умирать от яда (между прочим, это реальный случай — [12])? Ни Макдональд, ни Крейтман нам тут не помогут, поскольку высокий dN/dS может быть и у гена, который быстро проэволюционировал миллион лет назад, и ничего не знает про приспособления к яду. Зато поможет рекомбинация.
При мейозе в геноме происходит рекомбинация — гомологичные хромосомы обмениваются кусочками. Гомологичные хромосомы — это почти одинаковые хромосомы, содержащие одинаковые гены, но в немного разных вариантах. Одна из них организму достается от матери, а другая — от отца. Если мы рассмотрим две мутации, находящиеся на одной хромосоме на каком-то расстоянии друг от друга, рекомбинация может их расцепить — получится хромосома, на которой есть только одна из этих мутаций. Чем ближе друг к другу находятся эти мутации, тем меньше вероятность того, что рекомбинация их расцепит, и тем больше в среднем для этого понадобится времени (поколений) (рис. 5).
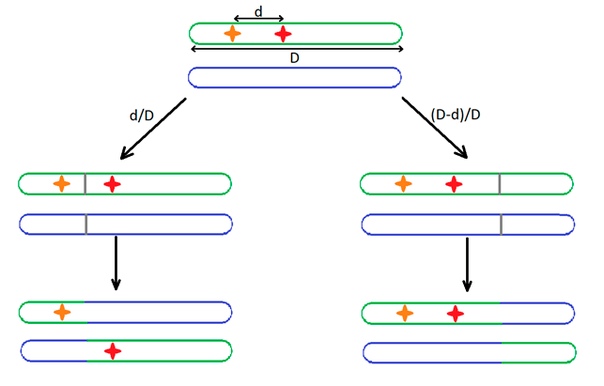
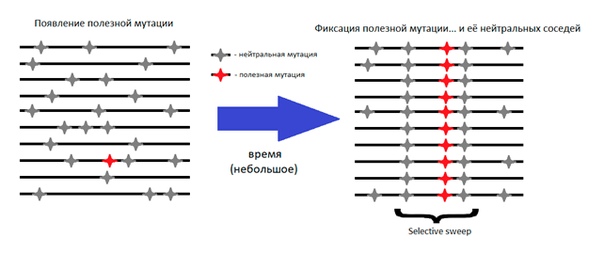
нова воспользуемся тем фактом, что положительный отбор фиксирует полезную мутацию быстро. За это время рекомбинация вряд ли успеет произойти где-нибудь неподалеку от этой мутации. Сформулируем более строго — чем более близкую позицию мы рассматриваем, тем меньше вероятность того, что между ней и интересующей нас мутацией произошла рекомбинация. Поэтому полезная мутация, закрепляясь, «потащит» за собой все окрестные мутации (нейтральные и даже слабовредные), и в месте, где происходил недавний положительный отбор, будет заметно локальное понижение нуклеотидного разнообразия в геноме — этот участок будет одинаковым у всей популяции [13]. Конечно, с течением времени там будут накапливаться мутации, и оно вернется к нормальному уровню. Но ведь это нам и на руку — если участок с пониженным разнообразием заметен, значит, с момента действия отбора времени прошло очень немного. Явление понижения нуклеотидного разнообразия вокруг положительно отбираемой мутации называется selective sweep, что означает «выметание» полиморфизмов из популяции (рис. 6).
Так как же это, черт побери, поможет в лечении рака!?
Ладно-ладно. Сейчас будет.
Раковую опухоль можно рассматривать как популяцию одноклеточных организмов в нашем теле. Успешными будут считаться те из них, которые размножаются и распространяются по телу человека быстрее других. Соответственно, для раковых клеток полезными, то есть поддерживаемыми положительным отбором, будут те мутации, которые помогают им в распространении и размножении. Было бы неплохо знать, какие же это, собственно, мутации. Тогда, например, можно будет предсказывать развитие рака.
Описанный в предыдущем разделе прием с раком не пройдет, потому что он опирается на процесс, связанный с рекомбинацией. А рекомбинация происходит только при половом размножении. Но вот незадача — полового процесса у раковых клеток нет. Поэтому когда в одной из них появляется полезная мутация, в популяции (то есть в опухоли) распространяются клоны этой клетки, неся в себе весь ее геном. А в геноме, конечно, была не только эта полезная мутация, но и куча нейтральных и, возможно, слабовредных. Ну и как теперь понять, какая из них полезная?
Но представьте, что можно было бы запускать процесс адаптации с самого начала вновь и вновь и наблюдать, как раз за разом в популяции распространяются некие клоны. Как тогда можно было бы понять, какие мутации в них полезны? Очевидно, те, которые раз за разом присутствуют в этих клонах! Ведь нейтральным мутациям в распространяющихся клонах просто повезло находиться в одном геноме с полезной мутацией. Но если мутация всегда находится в успешном клоне — списать ее успех на удачу становится сложно.
Так вот: процесс адаптации раковых клеток не просто можно запускать вновь и вновь — именно это и происходит, когда мы обследуем новых пациентов. Если у каждого из них клетки содержат одну и ту же мутацию (такие мутации называются рекуррентными), видимо, она полезна для раковой опухоли, и поддерживается положительным отбором [14]. Вот так все просто.
Повторюсь, положительно отбираемые в раковой опухоли гены — это как раз те гены, поломки в которых вызывают эту опухоль и позволяют ей расти. Если узнать, что это за гены, можно, во-первых, предсказывать риск развития рака у пациента, посмотрев ему в эти гены, а во-вторых — сконцентрироваться на их изучении и понять, что именно в них сломалось и почему они вызывают рост опухоли. Знание механизма позволяет разработать лекарственный препарат — а это уже намного приятнее, чем простое предсказание риска!
Балансирующий отбор
Балансирующий отбор — такой, который не ведет к закреплению мутаций, а поддерживает их численность на промежуточном уровне. Поэтому в участке с балансирующим отбором будет наблюдаться полиморфизм.
До 70-х годов XX века балансирующий отбор был почитаем и любим массами популяционных генетиков [15]. Успех его был вызван тем, что ему приписывали поддержание полиморфизмов в популяциях [16], которых с течением времени находили все больше. Однако в конце 70-х Мотоо Кимура убедил всех (или почти всех), что полиморфизмы в большинстве своем нейтральны [17]. Они — дрейф генов, который можно наблюдать в реальном времени. И красивых моделей про балансирующий отбор стало гораздо меньше. По-видимому, ситуация останется такой, пока кому-нибудь не удастся показать, что в геномах живых организмов (хотя бы каких-нибудь!) балансирующий отбор играет важную роль.
Доказанных случаев действия балансирующего отбора, как можно понять из предыдущего абзаца, немного. Самые популярные из них — это гены MHC у человека [18] (major histocompatibility complex — главный комплекс гистосовместимости) и мутация в гемоглобине, вызывающая серповидно-клеточную анемию [19].
Я, однако, расскажу вкратце о некоторых моделях балансирующего отбора. Мне кажется, это занятное упражнение — подумать, какие силы могли бы действовать так заковыристо, что ни одна из мутаций в данном участке не могла бы считаться во всех отношениях полезной.
- Частотно-зависимый отбор. Я упоминал о нем в начале. Если мутация считается полезной только пока она редкая, она не закрепится (потому что это будет значить, что она частая и, следовательно, вредная), но и не исчезнет (потому что, исчезая, она будет становиться редкой, и, следовательно, полезной).
- Преимущество гетерозигот. Предположим, по какому-то гену гомозиготы (носители одинаковых вариантов гена) имеют пониженную приспособленность по отношению к гетерозиготам (носителям разных вариантов гена). Соответственно, если мутация будет близка к закреплению, все чаще она будет оказываться в гомозиготе, и ее носители будут иметь низкую приспособленность. Наоборот, когда мутация будет близка к исчезновению, она чаще станет оказываться в гетерозиготе, и ее носители буду иметь высокую приспособленность.
- Пространственная/временная неоднородность. Если популяция живет в неоднородной среде, в некоторых областях местообитания могут быть полезны одни мутации, а в других — другие. Если члены популяции свободно перемещаются между этими частями, из поколения в поколение одна и та же мутация будет то полезной, то вредной, и будет то увеличиваться в частоте, то уменьшаться. Аналогично во времени — если сегодня полезна одна мутация, а завтра — другая, каждая мутация будет то вредной, то полезной, и опять не закрепится и не исчезнет.
- Множественный эффект гена. Например, мутация может быть полезной у самцов и вредной у самок. Поскольку каждая мутация с одинаковой вероятностью появляется у самцов и у самок, половину своего существования она будет полезной, а половину — вредной, поэтому не будет закрепляться или исчезать.
Как видите, довольно разные процессы могут приводить к одному и тому же эффекту. Но на самом деле, при поиске балансирующего отбора в геноме не имеет значения, каков был механизм его возникновения.
Я упомяну только один, наверное, самый популярный способ. Про другие можно почитать статью [20]. И способ, на самом деле, очень простой. Нужно опять обратиться к полиморфизмам в популяции.
Если я вижу в популяции полиморфизм, он может говорить об одном из трех случаев:
- Я вижу слабовредную мутацию. Ничего страшного, скоро я ее не увижу — отбор об этом позаботится.
- Я вижу нейтральную мутацию. Рано или поздно под действием генетического дрейфа она закрепится или исчезнет.
- Я вижу балансирующий отбор. В стабильных условиях полиморфизм потенциально может сохраняться вечно... Но условия никогда не бывают стабильными, поэтому, конечно, что-то с этим полиморфизмом когда-нибудь произойдет.
Можно заметить, что только балансирующий отбор может объяснить долгое сохранение полиморфизма в популяции. И если бы мы, посмотрев в популяцию, могли каким-то образом понять, что наблюдаемому нами полиморфизму многие сотни тысяч лет — тут уж мы бы точно решили, что виной всему балансирующий отбор.
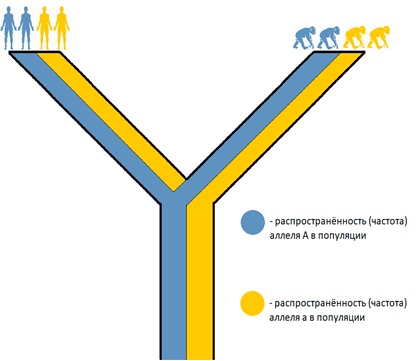
Но на самом деле можно понять, что полиморфизму многие сотни тысяч лет! Для этого всего лишь нужно посмотреть на то же самое место в геноме близкого вида. Если полиморфизм существовал до момента дивергенции этих видов (дивергенция — отделение одного вида от другого в процессе эволюции), он будет присутствовать и у него (рис. 7). Таким образом было доказано влияние балансирующего отбора на MHC [18].
Проблемы у такого метода есть. Конечно, да, если полиморфизм древний — он, скорее всего, вызван балансирующим отбором. Но ведь если полиморфизм молодой — это вовсе не значит, что он балансирующим отбором не вызван. Более того, случись так, что балансирующий отбор по природе своей действует только на протяжении короткого времени (что правдоподобно [21]), мы так никогда его не обнаружим! Да и в конце концов, после дивергенции виды, скорее всего, будут находиться в разных условиях — возможно, один из них окажется в таких, в которых условия для балансирующего отбора перестанут соблюдаться (один из аллелей станет безусловно вредным, а другой — безусловно полезным), и в этом виде полиморфизм исчезнет. И опять балансирующий отбор не будет замечен.
Из-за таких вот моментов доказательство балансирующего отбора очень сильно осложнено. Добавлю только, что недавний балансирующий отбор по своим побочным эффектам очень трудно различим с положительным отбором, поэтому искать его на коротких временных промежутках — тоже не самое приятное занятие [20].
Заключение. Ведь должно быть заключение
Кажется, это то самое место, где я должен выражать восторги относительно развития современной науки и надежды на скорейшее избавление человечества от страшных недугов. По тематике статьи выглядит так, что наиболее уместно здесь упомянуть рак — мол, вот, скоро эволюционная геномика (между прочим, так называется наука, которая всем этим занимается) идентифицирует нам все положительно отбираемые в опухолях варианты генов, и мы начнем за ними яростно следить и предотвращать развитие рака. Звучит неплохо. И даже, возможно, правдиво [10]. Но, честно говоря, я не люблю такие заключения.
Если нужно обязательно что-нибудь заключить, вот, что я заключу. Примерно то же, с чего начал, на самом деле. Сейчас, в геномную эру, быть эволюционным биологом хорошо и приятно: открывается море возможностей для изучения эволюционных процессов на уровне генов, и для этого доступно множество методов, о некоторых из которых я рассказал. Нет, не то чтобы все проблемы решены — вспомните тот же балансирующий отбор. Но когда у вас в каждом рукаве по 36 тузов, играть становится намного проще (если только вы не играете в кости).
Литература
- Laurence M Cook. (2003). The Rise and Fall of the Carbonaria Form of the Peppered Moth. The Quarterly Review of Biology. 78, 399-417;
- Шмальгаузен И.И. Факторы эволюции (теория стабилизирующего отбора). М.: «Наука», 1968. — 452 с.;
- A. E. Vinogradov, O. V. Anatskaya. (2017). DNA helix: the importance of being AT-rich. Mamm Genome. 28, 455-464;
- Как нонсенс-мутации ген обижали, и что потом было;
- Meneely Ph., Hoang R.D., Okeke I.N., Heston K. (2017). Genetics: Genes, Genomes, and Evolution. Oxford University Press, 2017. — 776 p.;
- Rasmus Nielsen, Carlos Bustamante, Andrew G Clark, Stephen Glanowski, Timothy B Sackton, et. al.. (2005). A Scan for Positively Selected Genes in the Genomes of Humans and Chimpanzees. PLoS Biol. 3, e170;
- E. J. Vallender. (2004). Positive selection on the human genome. Human Molecular Genetics. 13, R245-R254;
- John H. McDonald, Martin Kreitman. (1991). Adaptive protein evolution at the Adh locus in Drosophila. Nature. 351, 652-654;
- Mark P. Zwart, Martijn F. Schenk, Sungmin Hwang, Bertha Koopmanschap, Niek de Lange, et. al.. (2018). Unraveling the causes of adaptive benefits of synonymous mutations in TEM-1 ?-lactamase. Heredity. 121, 406-421;
- Samuel F. Bakhoum, Dan A. Landau. (2017). Cancer Evolution: No Room for Negative Selection. Cell. 171, 987-989;
- Zhang J. (2003). Evolution of the Human ASPM Gene, a Major Determinant of Brain Size. Genetics. 4, 2063–2070;
- M. H. Kohn, H.-J. Pelz, R. K. Wayne. (2000). Natural selection mapping of the warfarin-resistance gene. Proceedings of the National Academy of Sciences. 97, 7911-7915;
- JOHN MAYNARD, JOHN HAIGH. (2007). The hitch-hiking effect of a favourable gene. Genet. Res.. 89, 391-403;
- I?igo Martincorena, Keiran M. Raine, Moritz Gerstung, Kevin J. Dawson, Kerstin Haase, et. al.. (2017). Universal Patterns of Selection in Cancer and Somatic Tissues. Cell. 171, 1029-1041.e21;
- Andrew D. Gloss, Noah K. Whiteman. (2016). Balancing Selection: Walking a Tightrope. Current Biology. 26, R73-R76;
- Dobzhansky T. Genetics of the evolutionary process. Columbia University Press, 1970. — 505 p.;
- Kimura M. The Neutral Theory of Molecular Evolution. Cambridge University Press, 1983. — 387 p.;
- Weimin Fan, Masanori Kasahara, Jutta Gutknecht, Dagmar Klein, Werner E. Mayer, et. al.. (1989). Shared class II MHC polymorphisms between humans and chimpanzees. Human Immunology. 26, 107-121;
- Michael Aidoo, Dianne J Terlouw, Margarette S Kolczak, Peter D McElroy, Feiko O ter Kuile, et. al.. (2002). Protective effects of the sickle cell gene against malaria morbidity and mortality. The Lancet. 359, 1311-1312;
- Anna Fijarczyk, Wies?aw Babik. (2015). Detecting balancing selection in genomes: limits and prospects. Mol Ecol. 24, 3529-3545;
- Saurabh Asthana, Steffen Schmidt, Shamil Sunyaev. (2005). A limited role for balancing selection. Trends in Genetics. 21, 30-32.
Источник: m.vk.com