Глубокое обучение с R и Keras на примере Carvana Image Masking Challenge
МЕНЮ
Искусственный интеллект
Поиск
Регистрация на сайте
Помощь проекту
ТЕМЫ
Новости ИИ
Голосовой помощник
Городские сумасшедшие
ИИ в медицине
ИИ проекты
Искусственные нейросети
Слежка за людьми
Угроза ИИ
Компьютерные науки
Машинное обуч. (Ошибки)
Машинное обучение
Машинный перевод
Реализация ИИ
Реализация нейросетей
Создание беспилотных авто
Трезво про ИИ
Философия ИИ
Генетические алгоритмы
Капсульные нейросети
Основы нейронных сетей
Распознавание лиц
Распознавание образов
Распознавание речи
Техническое зрение
Чат-боты
Авторизация
2017-10-30 19:47
 Привет, Хабр!
Привет, Хабр!Пользователи R долгое время были лишены возможности приобщиться к deep learning-у, оставаясь в рамках одного языка программирования. С выходом MXNet ситуация стала меняться, но своеобразная документация и частые изменения, ломающие обратную совместимость, все еще ограничивают популярность данной библиотеки.
Гораздо привлекательнее выглядит использование R-интерфейсов к TensorFlow и Keras с бекендами на выбор (TensorFlow, Theano, CNTK), подробной документацией и множеством примеров. В этом сообщении будет разобрано решение задачи сегментации изображений на примере соревнования Carvana Image Masking Challenge (победители), в котором требуется научиться отделять автомобили, сфотографированные с 16 разных ракурсов, от фона. "Нейросетевая" часть полностью реализована на Keras, за обработку изображений отвечает magick (интерфейс к ImageMagick), параллельная обработка обеспечивается parallel+doParallel+foreach (Windows) или parallel+doMC+foreach (Linux).
Содержание:
- Установка всего необходимого
- Работа с изображениями: magick как альтернатива OpenCV
- Параллельное выполнение кода на R в ОС Windows и Linux
- reticulate и итераторы
- Задача сегментации и функция потерь для нее
- Архитектура U-Net
- Обучение модели
- Предсказания на основе модели
1. Установка всего необходимого
Будем считать, что у читателя уже есть GPU от Nvidia с ?4 Гб памяти (можно и меньше, но будет не так интересно), а также установлены библиотеки CUDA и cuDNN. Для Linux установка последних происходит просто (одно из многочисленных руководства), а для Windows — еще проще (см. раздел "CUDA & cuDNN" руководства).
Далее желательно установить дистрибутив Anaconda с Python 3; для экономии места можно выбрать минимальный вариант Miniconda. Если вдруг версия Python в дистрибутиве опережает последнюю поддерживаемую со стороны Tensorflow версию, заменить ее можно командой вида conda install python=3.6. Также все будет работать с обычным Python-ом и виртуальными окружениями.
Список используемых R-пакетов выглядит следующим образом:
Список пакетов для Windows
library(keras) library(magick) library(abind) library(reticulate) library(parallel) library(doParallel) library(foreach)Список пакетов для Linux
library(keras) library(magick) library(abind) library(reticulate) library(parallel) library(doMC) library(foreach)Все они устанавливаются с CRAN, но Keras лучше брать с Github: devtools::install_github("rstudio/keras"). Последующий запуск команды install_keras() создаст conda-окружение и установит в нем правильные версии Python-овских Tensorflow и Keras. Если эта команда по какой-то причине отказалась корректно работать (например, не смогла найти нужный дистрибутив Python), или же требуются специфические версии используемых библиотек, следует создать conda-окружение самому, установить в нем нужные пакеты, а затем в R указать пакету reticulate это окружение командой use_condaenv().
Список используемых далее параметров:
input_size <- 128 # Ширина и высота изображений, подаваемых на вход нейросети epochs <- 30 # Число эпох batch_size <- 16 # Размер батча orig_width <- 1918 # Ширина исходных изображений orig_height <- 1280 # Высота исходных изображений train_samples <- 5088 # Размер обучающей выборки train_index <- sample(1:train_samples, round(train_samples * 0.8)) # 80% val_index <- c(1:train_samples)[-train_index] # Папки с картинками images_dir <- "input/train/" masks_dir <- "input/train_masks/"2. Работа с изображениями: magick как альтернатива OpenCV
При решении задач машинного обучения на графических данных нужно уметь, как минимум, читать изображения с диска и передавать их в нейросетку в виде массивов. Обычно требуется также уметь выполнять разнообразные трансформации изображений, чтобы реализовать так называемую аугментацию — дополнение обучающей выборки искусственными примерами, созданными из фактически присутствующих в самой обучающей выборке образцов. Аугментация (почти) всегда способна дать прирост качества модели: базовое понимание можно получить, например, из этого сообщения. Забегая вперед, отметим, что все это нужно делать быстро и многопоточно: даже на относительно быстром CPU и относительно медленной видеокарте подготовительный этап может оказаться более ресурсоемким, чем собственно обучение нейросети.
В Python для работы с изображениями традиционно используется OpenCV. Версии этой мегабиблиотеки для R пока не создали, вызов ее функций посредством reticulate выглядит неспортивным решением, поэтому будем выбирать из имеющихся альтернатив.
Топ-3 самых мощных графических пакетов выглядит следующим образом:
EBImage — пакет создан с использованием S4-классов и размещен в репозитории Bioconductora-а, что подразумевает самые высокие требования к качеству как самого пакета, так и его документации. К сожалению, насладиться обширными возможностями данного программного продукта мешает его крайне низкая скорость работы.
imager — этот пакет выглядит поинтереснее в плане производительности, поскольку основную работу в нем выполняет скомпилированный код в лице сишной библиотеки CImg. Среди достоинств можно отметить поддержку "конвейерного" оператора
%>%(и других операторов из magrittr), тесную интеграцию с пакетами из т.н. tidyverse, включая ggplot2, а также поддержку идеологии split-apply-combine. И лишь непонятный баг, делающий неработоспособными функции для чтения картинок на некоторых ПК, помешал автору этого сообщения остановить свой выбор на данном пакете.- magick — оболочка для ImageMagick, разработанный и активно развиваемый участниками сообщества rOpenSci. Сочетает в себе все плюсы предыдущего пакета, стабильность, безглючность и киллер-фичу (бесполезную в рамках нашей задачи) в виде интеграции с OCR-библиотекой Tesseract. Замеры скорости при выполнении чтения и трансформации картинок на разном числе ядер приведены ниже. Из минусов можно отметить местами эзотерический синтаксис: например, для обрезки или изменения размера нужно передать функции строку вида
"100x150+50"вместо привычных аргументов типаheightиwigth. А поскольку наши вспомогательные функции для препроцессинга будут параметризованы как раз по этим величинам, придется использовать некрасивые конструкцииpaste0(...)илиsprintf(...).
Здесь и далее мы будем в общих чертах воспроизводить решение Kaggle Carvana Image Masking Challenge solution with Keras от Peter-а Giannakopoulos-а.
Читать файлы нужно парами — изображение и соответствующую ему маску, также к изображению и маске нужно применять одинаковые преобразования (повороты, сдвиги, отражения, изменения масштаба) при использовании аугментации. Реализуем чтение в виде одной функции, которая сразу же будет уменьшать картинки под нужный размер:
imagesRead <- function(image_file, mask_file, target_width = 128, target_height = 128) { img <- image_read(image_file) img <- image_scale(img, paste0(target_width, "x", target_height, "!")) mask <- image_read(mask_file) mask <- image_scale(mask, paste0(target_width, "x", target_height, "!")) list(img = img, mask = mask) }Результат работы функции с наложением маски на изображение:
img <- "input/train/0cdf5b5d0ce1_01.jpg" mask <- "input/train_masks/0cdf5b5d0ce1_01_mask.png" x_y_imgs <- imagesRead(img, mask, target_width = 400, target_height = 400) image_composite(x_y_imgs$img, x_y_imgs$mask, operator = "blend", compose_args = "60") %>% image_write(path = "pics/pic1.jpg", format = "jpg")
Первым видом аугментации будет изменение яркости (brightness), насыщенности (saturation) и тона (hue). По понятным причинам применяется она к цветному изображению, но не к черно-белой маске:
randomBSH <- function(img, u = 0, brightness_shift_lim = c(90, 110), # percentage saturation_shift_lim = c(95, 105), # of current value hue_shift_lim = c(80, 120)) { if (rnorm(1) < u) return(img) brightness_shift <- runif(1, brightness_shift_lim[1], brightness_shift_lim[2]) saturation_shift <- runif(1, saturation_shift_lim[1], saturation_shift_lim[2]) hue_shift <- runif(1, hue_shift_lim[1], hue_shift_lim[2]) img <- image_modulate(img, brightness = brightness_shift, saturation = saturation_shift, hue = hue_shift) img }Это преобразование применяется с вероятностью 50% (в половине случаев будет возвращено исходное изображение:if (rnorm(1) < u) return(img)), величина изменения каждого из трех параметров выбирается случайным образом в пределах диапазона значений, заданного в процентах от исходной величины.
Также с вероятностью 50% будем использовать горизонтальные отражения изображения и маски:
randomHorizontalFlip <- function(img, mask, u = 0) { if (rnorm(1) < u) return(list(img = img, mask = mask)) list(img = image_flop(img), mask = image_flop(mask)) }Результат:
img <- "input/train/0cdf5b5d0ce1_01.jpg" mask <- "input/train_masks/0cdf5b5d0ce1_01_mask.png" x_y_imgs <- imagesRead(img, mask, target_width = 400, target_height = 400) x_y_imgs$img <- randomBSH(x_y_imgs$img) x_y_imgs <- randomHorizontalFlip(x_y_imgs$img, x_y_imgs$mask) image_composite(x_y_imgs$img, x_y_imgs$mask, operator = "blend", compose_args = "60") %>% image_write(path = "pics/pic2.jpg", format = "jpg")
Остальные преобразования для дальнейшего изложения не принципиальны, поэтому на них останавливаться не будем.
Последний этап — превращение картинок в массивы:
img2arr <- function(image, target_width = 128, target_height = 128) { result <- aperm(as.numeric(image[[1]])[, , 1:3], c(2, 1, 3)) # transpose dim(result) <- c(1, target_width, target_height, 3) return(result) } mask2arr <- function(mask, target_width = 128, target_height = 128) { result <- t(as.numeric(mask[[1]])[, , 1]) # transpose dim(result) <- c(1, target_width, target_height, 1) return(result) }Транспонирование нужно для того, чтобы строки изображения оставались строками в матрице: изображение формируется построчно (как движется луч развертки в кинескопе), в то время как матрицы в R заполняются по столбцам (column-major, или Fortran-style; для сравнения, в numpy можно переключаться между column-major и row-major форматами). Можно обойтись и без него, но так понятнее.
3. Параллельное выполнение кода на R в ОС Windows и Linux
Общее представление о параллельных вычислениях в R можно получить из руководств Package ‘parallel’, Getting Started with doParallel and foreach и Getting Started with doMC and foreach. Алгоритм работы следующий:
Запускаем кластер с нужным числом ядер:
cl <- makePSOCKcluster(4) # doParallelSOCK-кластеры являются универсальным решением, позволяющим в том числе использовать CPU нескольких ПК. К сожалению, наш пример с итераторами и обучением нейронной сети работает под Windows, но отказывается работать под Linux. В Linux можно воспользоваться альтернативным пакетом doMC, который создает кластеры с использованием форков исходного процесса. Остальные шаги выполнять не нужно:
registerDoMC(4) # doMCИ doParallel, и doMC служат посредниками между функциональностью parallel и foreach.
При использовании makePSOCKcluster() нужно подгрузить внутрь кластера необходимые пакеты и функции:
Загрузка пакетов и функций
clusterEvalQ(cl, { library(magick) library(abind) library(reticulate) imagesRead <- function(image_file, mask_file, target_width = 128, target_height = 128) { img <- image_read(image_file) img <- image_scale(img, paste0(target_width, "x", target_height, "!")) mask <- image_read(mask_file) mask <- image_scale(mask, paste0(target_width, "x", target_height, "!")) return(list(img = img, mask = mask)) } randomBSH <- function(img, u = 0, brightness_shift_lim = c(90, 110), # percentage saturation_shift_lim = c(95, 105), # of current value hue_shift_lim = c(80, 120)) { if (rnorm(1) < u) return(img) brightness_shift <- runif(1, brightness_shift_lim[1], brightness_shift_lim[2]) saturation_shift <- runif(1, saturation_shift_lim[1], saturation_shift_lim[2]) hue_shift <- runif(1, hue_shift_lim[1], hue_shift_lim[2]) img <- image_modulate(img, brightness = brightness_shift, saturation = saturation_shift, hue = hue_shift) img } randomHorizontalFlip <- function(img, mask, u = 0) { if (rnorm(1) < u) return(list(img = img, mask = mask)) list(img = image_flop(img), mask = image_flop(mask)) } img2arr <- function(image, target_width = 128, target_height = 128) { result <- aperm(as.numeric(image[[1]])[, , 1:3], c(2, 1, 3)) # transpose dim(result) <- c(1, target_width, target_height, 3) return(result) } mask2arr <- function(mask, target_width = 128, target_height = 128) { result <- t(as.numeric(mask[[1]])[, , 1]) # transpose dim(result) <- c(1, target_width, target_height, 1) return(result) } })Регистрируем кластер в качестве параллельного бекенда для foreach:
registerDoParallel(cl)После этого можно запускать код в параллельном режиме:
imgs <- list.files("input/train/", pattern = ".jpg", full.names = TRUE)[1:16] masks <- list.files("input/train_masks/", pattern = ".png", full.names = TRUE)[1:16] x_y_batch <- foreach(i = 1:16) %dopar% { x_y_imgs <- imagesRead(image_file = batch_images_list[i], mask_file = batch_masks_list[i]) # augmentation x_y_imgs$img <- randomBSH(x_y_imgs$img) x_y_imgs <- randomHorizontalFlip(x_y_imgs$img, x_y_imgs$mask) # return as arrays x_y_arr <- list(x = img2arr(x_y_imgs$img), y = mask2arr(x_y_imgs$mask)) } str(x_y_batch) # List of 16 # $ :List of 2 # ..$ x: num [1, 1:128, 1:128, 1:3] 0.953 0.957 0.953 0.949 0.949 ... # ..$ y: num [1, 1:128, 1:128, 1] 0 0 0 0 0 0 0 0 0 0 ... # $ :List of 2 # ..$ x: num [1, 1:128, 1:128, 1:3] 0.949 0.957 0.953 0.949 0.949 ... # ..$ y: num [1, 1:128, 1:128, 1] 0 0 0 0 0 0 0 0 0 0 ... # ....В конце не забываем остановить кластер:
stopCluster(cl)При помощи пакета microbenchmark проверим, каков выигрыш от использования нескольких ядер/потоков. На GPU c 4 Гб памяти можно работать с батчами по 16 пар изображений, значит, целесообразно использовать 2, 4, 8 или 16 потоков (время указано в секундах):
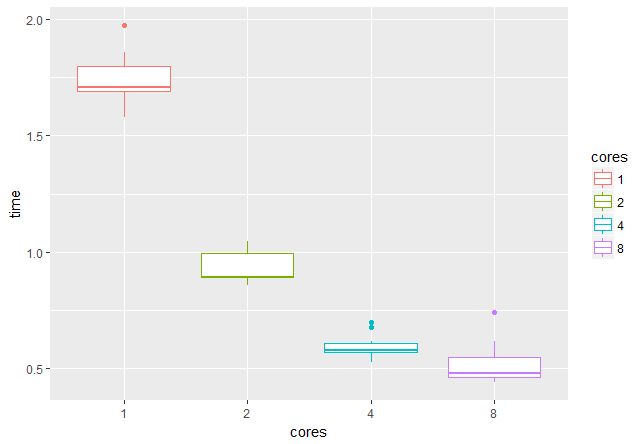
На 16 потоках проверить не было возможности, но видно, что при переходе от 1 к 4 потокам скорость возрастает примерно втрое, что очень радует.
4. reticulate и итераторы
Чтобы работать с данными, не помещающимися в памяти, воспользуемся итераторами из пакета reticulate. Основой является обычная функция-замыкание (closure), т.е.функция, при вызове возвращающая другую функцию вместе с окружением вызова:
train_generator
train_generator <- function(images_dir, samples_index, masks_dir, batch_size) { images_iter <- list.files(images_dir, pattern = ".jpg", full.names = TRUE)[samples_index] # for current epoch images_all <- list.files(images_dir, pattern = ".jpg", full.names = TRUE)[samples_index] # for next epoch masks_iter <- list.files(masks_dir, pattern = ".gif", full.names = TRUE)[samples_index] # for current epoch masks_all <- list.files(masks_dir, pattern = ".gif", full.names = TRUE)[samples_index] # for next epoch function() { # start new epoch if (length(images_iter) < batch_size) { images_iter <<- images_all masks_iter <<- masks_all } batch_ind <- sample(1:length(images_iter), batch_size) batch_images_list <- images_iter[batch_ind] images_iter <<- images_iter[-batch_ind] batch_masks_list <- masks_iter[batch_ind] masks_iter <<- masks_iter[-batch_ind] x_y_batch <- foreach(i = 1:batch_size) %dopar% { x_y_imgs <- imagesRead(image_file = batch_images_list[i], mask_file = batch_masks_list[i]) # augmentation x_y_imgs$img <- randomBSH(x_y_imgs$img) x_y_imgs <- randomHorizontalFlip(x_y_imgs$img, x_y_imgs$mask) # return as arrays x_y_arr <- list(x = img2arr(x_y_imgs$img), y = mask2arr(x_y_imgs$mask)) } x_y_batch <- purrr::transpose(x_y_batch) x_batch <- do.call(abind, c(x_y_batch$x, list(along = 1))) y_batch <- do.call(abind, c(x_y_batch$y, list(along = 1))) result <- list(keras_array(x_batch), keras_array(y_batch)) return(result) } }val_generator
val_generator <- function(images_dir, samples_index, masks_dir, batch_size) { images_iter <- list.files(images_dir, pattern = ".jpg", full.names = TRUE)[samples_index] # for current epoch images_all <- list.files(images_dir, pattern = ".jpg", full.names = TRUE)[samples_index] # for next epoch masks_iter <- list.files(masks_dir, pattern = ".gif", full.names = TRUE)[samples_index] # for current epoch masks_all <- list.files(masks_dir, pattern = "gif", full.names = TRUE)[samples_index] # for next epoch function() { # start new epoch if (length(images_iter) < batch_size) { images_iter <<- images_all masks_iter <<- masks_all } batch_ind <- sample(1:length(images_iter), batch_size) batch_images_list <- images_iter[batch_ind] images_iter <<- images_iter[-batch_ind] batch_masks_list <- masks_iter[batch_ind] masks_iter <<- masks_iter[-batch_ind] x_y_batch <- foreach(i = 1:batch_size) %dopar% { x_y_imgs <- imagesRead(image_file = batch_images_list[i], mask_file = batch_masks_list[i]) # without augmentation # return as arrays x_y_arr <- list(x = img2arr(x_y_imgs$img), y = mask2arr(x_y_imgs$mask)) } x_y_batch <- purrr::transpose(x_y_batch) x_batch <- do.call(abind, c(x_y_batch$x, list(along = 1))) y_batch <- do.call(abind, c(x_y_batch$y, list(along = 1))) result <- list(keras_array(x_batch), keras_array(y_batch)) return(result) } }Здесь в окружении вызова хранятся уменьшающиеся в ходе каждой эпохи списки обрабатываемых файлов, а также копии полных списков, которые используется в начале каждой следующей эпохи. В данной реализации не нужно беспокоиться о случайном перемешивании файлов — каждый батч получается путем случайной выборки.
Как было показано выше, x_y_batch представляет собой список из 16 списков, каждый из которых является списком из 2 массивов. Функция purrr::transpose() выворачивает вложенные списки наизнанку, и мы получаем список из 2 списков, каждый из которых является списком из 16 массивов. abind() объединяет массивы вдоль указанного измерения, do.call() передает во внутреннюю функцию произвольное число аргументов. Дополнительные аргументы (along = 1) задаются весьма причудливым способом: do.call(abind, c(x_y_batch$x, list(along = 1))).
Осталось превратить эти функции в объекты, понятные для Keras, при помощи py_iterator():
train_iterator <- py_iterator(train_generator(images_dir = images_dir, masks_dir = masks_dir, samples_index = train_index, batch_size = batch_size)) val_iterator <- py_iterator(val_generator(images_dir = images_dir, masks_dir = masks_dir, samples_index = val_index, batch_size = batch_size))Вызов iter_next(train_iterator) вернет результат выполнения одной итерации, что полезно на этапе отладки.
5. Задача сегментации и функция потерь для нее
Задачу сегментации можно рассматривать как попиксельную классификацию: предсказывается принадлежность каждого пикселя к тому или иному классу. Для случая двух классов результат будет представлять собой маску; если классов больше двух, число масок будет равно числу классов минус 1 (аналог one-hot encodind). В нашем соревновании классов всего два (машина и фон), метрикой качества выступает dice-коэффициент. Рассчитывает он так:
K <- backend() dice_coef <- function(y_true, y_pred, smooth = 1.0) { y_true_f <- K$flatten(y_true) y_pred_f <- K$flatten(y_pred) intersection <- K$sum(y_true_f * y_pred_f) result <- (2 * intersection + smooth) / (K$sum(y_true_f) + K$sum(y_pred_f) + smooth) return(result) }Оптимизировать будем функцию потерь, являющуюся суммой перекрестной энтропии и 1 - dice_coef:
bce_dice_loss <- function(y_true, y_pred) { result <- loss_binary_crossentropy(y_true, y_pred) + (1 - dice_coef(y_true, y_pred)) return(result) }6. Архитектура U-Net
U-Net — классическая архитектура для решения задач сегментации. Принципиальная схема:
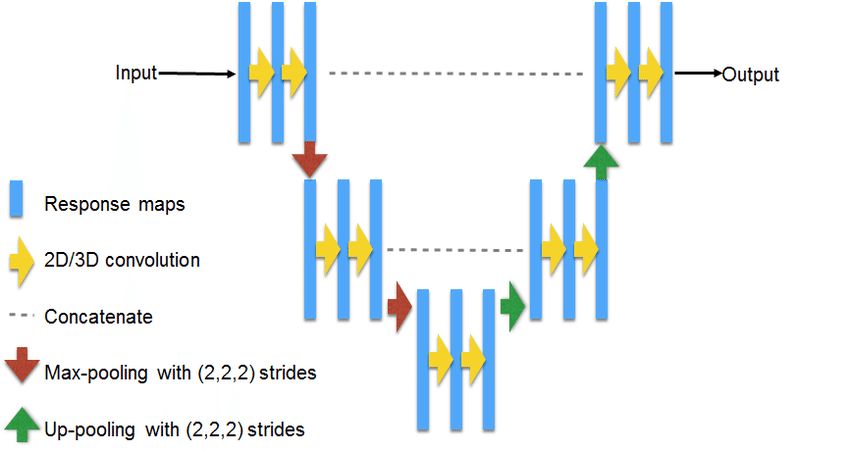
Источник: https://www.researchgate.net/figure/311715357_fig3_Fig-3-U-NET-Architecture
Реализация для картинок 128х128:
U-Net 128
get_unet_128 <- function(input_shape = c(128, 128, 3), num_classes = 1) { inputs <- layer_input(shape = input_shape) # 128 down1 <- inputs %>% layer_conv_2d(filters = 64, kernel_size = c(3, 3), padding = "same") %>% layer_batch_normalization() %>% layer_activation("relu") %>% layer_conv_2d(filters = 64, kernel_size = c(3, 3), padding = "same") %>% layer_batch_normalization() %>% layer_activation("relu") down1_pool <- down1 %>% layer_max_pooling_2d(pool_size = c(2, 2), strides = c(2, 2)) # 64 down2 <- down1_pool %>% layer_conv_2d(filters = 128, kernel_size = c(3, 3), padding = "same") %>% layer_batch_normalization() %>% layer_activation("relu") %>% layer_conv_2d(filters = 128, kernel_size = c(3, 3), padding = "same") %>% layer_batch_normalization() %>% layer_activation("relu") down2_pool <- down2 %>% layer_max_pooling_2d(pool_size = c(2, 2), strides = c(2, 2)) # 32 down3 <- down2_pool %>% layer_conv_2d(filters = 256, kernel_size = c(3, 3), padding = "same") %>% layer_batch_normalization() %>% layer_activation("relu") %>% layer_conv_2d(filters = 256, kernel_size = c(3, 3), padding = "same") %>% layer_batch_normalization() %>% layer_activation("relu") down3_pool <- down3 %>% layer_max_pooling_2d(pool_size = c(2, 2), strides = c(2, 2)) # 16 down4 <- down3_pool %>% layer_conv_2d(filters = 512, kernel_size = c(3, 3), padding = "same") %>% layer_batch_normalization() %>% layer_activation("relu") %>% layer_conv_2d(filters = 512, kernel_size = c(3, 3), padding = "same") %>% layer_batch_normalization() %>% layer_activation("relu") down4_pool <- down4 %>% layer_max_pooling_2d(pool_size = c(2, 2), strides = c(2, 2)) # 8 center <- down4_pool %>% layer_conv_2d(filters = 1024, kernel_size = c(3, 3), padding = "same") %>% layer_batch_normalization() %>% layer_activation("relu") %>% layer_conv_2d(filters = 1024, kernel_size = c(3, 3), padding = "same") %>% layer_batch_normalization() %>% layer_activation("relu") # center up4 <- center %>% layer_upsampling_2d(size = c(2, 2)) %>% {layer_concatenate(inputs = list(down4, .), axis = 3)} %>% layer_conv_2d(filters = 512, kernel_size = c(3, 3), padding = "same") %>% layer_batch_normalization() %>% layer_activation("relu") %>% layer_conv_2d(filters = 512, kernel_size = c(3, 3), padding = "same") %>% layer_batch_normalization() %>% layer_activation("relu") %>% layer_conv_2d(filters = 512, kernel_size = c(3, 3), padding = "same") %>% layer_batch_normalization() %>% layer_activation("relu") # 16 up3 <- up4 %>% layer_upsampling_2d(size = c(2, 2)) %>% {layer_concatenate(inputs = list(down3, .), axis = 3)} %>% layer_conv_2d(filters = 256, kernel_size = c(3, 3), padding = "same") %>% layer_batch_normalization() %>% layer_activation("relu") %>% layer_conv_2d(filters = 256, kernel_size = c(3, 3), padding = "same") %>% layer_batch_normalization() %>% layer_activation("relu") %>% layer_conv_2d(filters = 256, kernel_size = c(3, 3), padding = "same") %>% layer_batch_normalization() %>% layer_activation("relu") # 32 up2 <- up3 %>% layer_upsampling_2d(size = c(2, 2)) %>% {layer_concatenate(inputs = list(down2, .), axis = 3)} %>% layer_conv_2d(filters = 128, kernel_size = c(3, 3), padding = "same") %>% layer_batch_normalization() %>% layer_activation("relu") %>% layer_conv_2d(filters = 128, kernel_size = c(3, 3), padding = "same") %>% layer_batch_normalization() %>% layer_activation("relu") %>% layer_conv_2d(filters = 128, kernel_size = c(3, 3), padding = "same") %>% layer_batch_normalization() %>% layer_activation("relu") # 64 up1 <- up2 %>% layer_upsampling_2d(size = c(2, 2)) %>% {layer_concatenate(inputs = list(down1, .), axis = 3)} %>% layer_conv_2d(filters = 64, kernel_size = c(3, 3), padding = "same") %>% layer_batch_normalization() %>% layer_activation("relu") %>% layer_conv_2d(filters = 64, kernel_size = c(3, 3), padding = "same") %>% layer_batch_normalization() %>% layer_activation("relu") %>% layer_conv_2d(filters = 64, kernel_size = c(3, 3), padding = "same") %>% layer_batch_normalization() %>% layer_activation("relu") # 128 classify <- layer_conv_2d(up1, filters = num_classes, kernel_size = c(1, 1), activation = "sigmoid") model <- keras_model( inputs = inputs, outputs = classify ) model %>% compile( optimizer = optimizer_rmsprop(lr = 0.0001), loss = bce_dice_loss, metrics = c(dice_coef) ) return(model) } model <- get_unet_128()Фигурные скобки в {layer_concatenate(inputs = list(down4, .), axis = 3)} нужны для подстановки объекта в виде нужного аргумента, а не в виде первого по счету, как в противном случае делает оператор %>%. Можно предложить много модификаций этой архитектуры: использовать layer_conv_2d_transpose вместо layer_upsampling_2d, применить раздельные свертки layer_separable_conv_2d вместо обычных, поэкспериментировать с числов фильтров и с настройками оптимизаторов. По ссылке Kaggle Carvana Image Masking Challenge solution with Keras имеются варианты для разрешений вплоть до 1024х1024, которые так же легко портируются на R.
В нашей модели достаточно много параметров:
# Total params: 34,540,737 # Trainable params: 34,527,041 # Non-trainable params: 13,6967. Обучение модели
Тут все просто. Запускаем Tensorboard:
tensorboard("logs_r")В качестве альтернативы доступен пакет tfruns, который добавляет в IDE RStudio некий аналог Tensorboard и позволяет упорядочить работу по обучению нейросетей.
Указываем callback-и. Будем использовать раннюю остановку, уменьшать скорость обучения при выходе на плато и сохранять веса лучшей модели:
callbacks_list <- list( callback_tensorboard("logs_r"), callback_early_stopping(monitor = "val_python_function", min_delta = 1e-4, patience = 8, verbose = 1, mode = "max"), callback_reduce_lr_on_plateau(monitor = "val_python_function", factor = 0.1, patience = 4, verbose = 1, epsilon = 1e-4, mode = "max"), callback_model_checkpoint(filepath = "weights_r/unet128_{epoch:02d}.h5", monitor = "val_python_function", save_best_only = TRUE, save_weights_only = TRUE, mode = "max" ) )Запускаем обучение и ждем. На GTX 1050ti одна эпоха занимает порядка 10 минут:
model %>% fit_generator( train_iterator, steps_per_epoch = as.integer(length(train_index) / batch_size), epochs = epochs, validation_data = val_iterator, validation_steps = as.integer(length(val_index) / batch_size), verbose = 1, callbacks = callbacks_list )8. Предсказания на основе модели
Под спойлером приводится демо-версия кода для предсказания с использованием run-length encoding.
Предсказания на основе модели
test_dir <- "input/test/" test_samples <- 100064 test_index <- sample(1:test_samples, 1000) load_model_weights_hdf5(model, "weights_r/unet128_08.h5") # best model imageRead <- function(image_file, target_width = 128, target_height = 128) { img <- image_read(image_file) img <- image_scale(img, paste0(target_width, "x", target_height, "!")) } img2arr <- function(image, target_width = 128, target_height = 128) { result <- aperm(as.numeric(image[[1]])[, , 1:3], c(2, 1, 3)) # transpose dim(result) <- c(1, target_width, target_height, 3) return(result) } arr2img <- function(arr, target_width = 1918, target_height = 1280) { img <- image_read(arr) img <- image_scale(img, paste0(target_width, "x", target_height, "!")) } qrle <- function(mask) { img <- t(mask) dim(img) <- c(128, 128, 1) img <- arr2img(img) arr <- as.numeric(img[[1]])[, , 2] vect <- ifelse(as.vector(arr) >= 0.5, 1, 0) turnpoints <- c(vect, 0) - c(0, vect) starts <- which(turnpoints == 1) ends <- which(turnpoints == -1) paste(c(rbind(starts, ends - starts)), collapse = " ") } cl <- makePSOCKcluster(4) clusterEvalQ(cl, { library(magick) library(abind) library(reticulate) imageRead <- function(image_file, target_width = 128, target_height = 128) { img <- image_read(image_file) img <- image_scale(img, paste0(target_width, "x", target_height, "!")) } img2arr <- function(image, target_width = 128, target_height = 128) { result <- aperm(as.numeric(image[[1]])[, , 1:3], c(2, 1, 3)) # transpose dim(result) <- c(1, target_width, target_height, 3) return(result) } qrle <- function(mask) { img <- t(mask) dim(img) <- c(128, 128, 1) img <- arr2img(img) arr <- as.numeric(img[[1]])[, , 2] vect <- ifelse(as.vector(arr) >= 0.5, 1, 0) turnpoints <- c(vect, 0) - c(0, vect) starts <- which(turnpoints == 1) ends <- which(turnpoints == -1) paste(c(rbind(starts, ends - starts)), collapse = " ") } }) registerDoParallel(cl) test_generator <- function(images_dir, samples_index, batch_size) { images_iter <- list.files(images_dir, pattern = ".jpg", full.names = TRUE)[samples_index] function() { batch_ind <- sample(1:length(images_iter), batch_size) batch_images_list <- images_iter[batch_ind] images_iter <<- images_iter[-batch_ind] x_batch <- foreach(i = 1:batch_size) %dopar% { img <- imageRead(image_file = batch_images_list[i]) # return as array arr <- img2arr(img) } x_batch <- do.call(abind, c(x_batch, list(along = 1))) result <- list(keras_array(x_batch)) } } test_iterator <- py_iterator(test_generator(images_dir = test_dir, samples_index = test_index, batch_size = batch_size)) preds <- predict_generator(model, test_iterator, steps = 10) preds <- foreach(i = 1:160) %dopar% { result <- qrle(preds[i, , , ]) } preds <- do.call(rbind, preds)Ничего нового этот код не содержит, кроме функции qrle, которая выдает предсказания в требуемом организаторами соревнования формате (за нее спасибо skoffer-у):
Гифка со сравнением оригинальной и предсказанной маски:

Отсутствие мелких деталей объясняется использованием картинок низкого разрешения — всего 128х128. При работе в более высоком разрешении результат будет, разумеется, лучше.
При нехватке памяти можно делать предсказания порциями по несколько тысяч наблюдений, а затем сохранять их в один файл.
Итого
В этом сообщении было показано, что и сидя на R можно не отставать от модных тенденций и успешно обучать глубокие нейросетки. Причем даже ОС Windows не в силах этому помешать.
Продолжение, как обычно, следует.
Источник: habrahabr.ru