Нейронная сеть на Python в 15 строк кода для диагностики диабета
МЕНЮ
Искусственный интеллект
Поиск
Регистрация на сайте
Помощь проекту
ТЕМЫ
Новости ИИ
Голосовой помощник
Городские сумасшедшие
ИИ в медицине
ИИ проекты
Искусственные нейросети
Слежка за людьми
Угроза ИИ
Компьютерные науки
Машинное обуч. (Ошибки)
Машинное обучение
Машинный перевод
Реализация ИИ
Реализация нейросетей
Создание беспилотных авто
Трезво про ИИ
Философия ИИ
Генетические алгоритмы
Капсульные нейросети
Основы нейронных сетей
Распознавание лиц
Распознавание образов
Распознавание речи
Техническое зрение
Чат-боты
Авторизация
2018-12-21 12:00

В этой статье опишем как минимальным средствами может быть создана и обучена нейронная сеть при помощи Python и библиотеки Keras.
Сегодня мы рассмотрим задачу, связанную с применением анализа медицинских данных, а именно — диагностики риска заболевания сахарным диабетом на основе состояния больного. Для этого мы напишем простую нейронную сеть, которая решает важную практическую задачу.
Библиотека Keras представляет собой высокоуровневый интерфейс для создания нейронных сетей. Keras написан на Python и работает поверх таких более низкоуровневых решений, как TensorFlow, CNTK и Theano. За счет этого программный код получается не только мощным, но и крайне компактным.
Экспериментальные данные, взятые из медицинского архива, представляют собой файл со значениями, разделенными запятыми, в котором каждая строка соответствует одной анкете. Подробную информацию о данных из датасета вы можете прочитать здесь. В наборе экспериментальных данных, состоящих из анонимных записей имеется девять параметров. Последний из них, целевой, показывает, наблюдался ли у пациента сахарный диабет или нет (соответственно, 1 или 0). Восемь остальных параметров также имеют численные значения:
- Число беременностей (все пациенты из источника – женщины не моложе 21 года индийской народности пима).
- Концентрация глюкозы в плазме через 2 часа после введения в пероральном глюкозотолерантном тесте.
- Диастолическое артериальное давление (мм рт. ст.).
- Толщина кожной складки в районе трицепса (мм).
- Концентрация инсулина в сыворотке крови (мкЕд/мл).
- Индекс массы тела (вес в кг/(рост в м)^2).
- Функция, описывающая генетическую предрасположенность к диабету (diabetes pedegree).
- Возраст (годы).
Программный код и датасет также доступны на Github.
Библиотека Keras позволяет запускать нейронные сети с минимальным количеством операций. В качестве модели нейронной сети используется последовательная Sequental из модуля keras.models с заданием слоев keras.layers типа Dense.
Python
| 1 2 3 | fromkeras.models importSequential fromkeras.layers importDense importnumpy |
Для последующей воспроизводимости результатов зафиксируем генератор случайных чисел при помощи функции random.seed() из библиотеки numpy. Считаем данные из датасета:
Python
| 1 2 | numpy.random.seed(2) dataset=numpy.loadtxt("prima-indians-diabetes.csv",delimiter=",") |
Разделим данные на матрицу признаков X и вектор целевой переменной Y (последний столбец датасета):
Python
| 1 2 | X=dataset[:,0:8] Y=dataset[:,8] |
Создаем модель нейронной сети:
Python
| 1 | model=Sequential() |
Опишем структуру модели нейронной сети. Определим входной, выходной и скрытые слои. Наша нейронная сеть будет иметь плотную (Dense) структуру – каждый нейрон связан со всеми нейронами следующего слоя. Выходной слой будет состоять из единственного нейрона, определяющего вероятность заболевания диабетом.
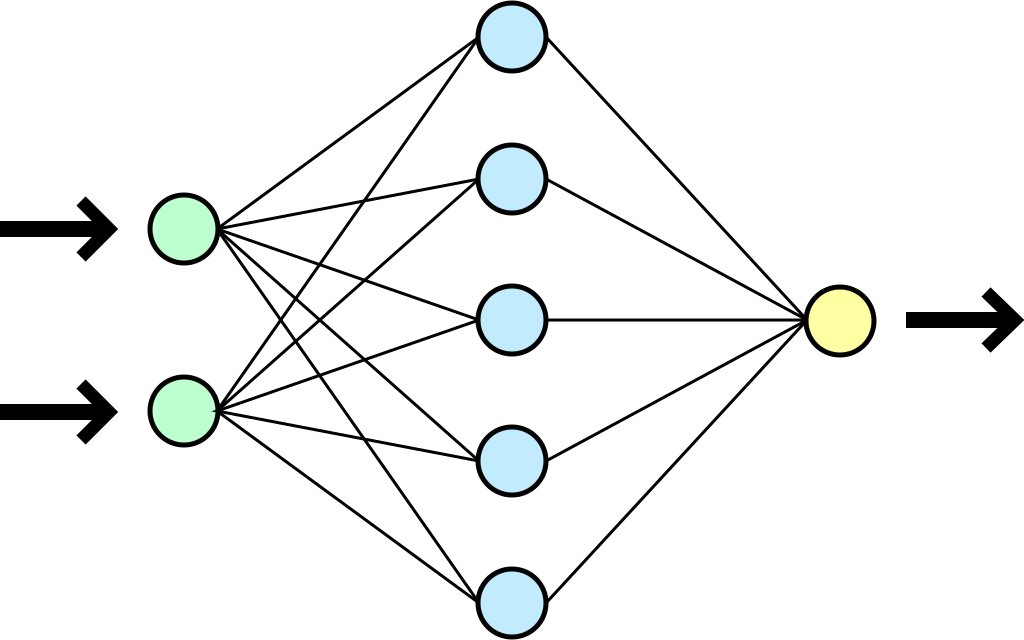
Слой добавляется к модели методом add(). Для входного слоя необходимо указать число признаков input_dim, равное в нашем случае 8:
Python
| 1 | model.add(Dense(12,input_dim=8,activation='relu')) |
Если наборы признаков образуют многомерную таблицу, то вместо параметра input_dim можно использовать параметр input_shape, принимающий кортеж с количеством элементов в каждом из измерений.
В качестве функции активации для всех слоев, кроме выходного, будем использовать функцию ReLU. Для выходного слоя воспользуемся сигмоидной функцией для определения конечной вероятности риска заболевания.
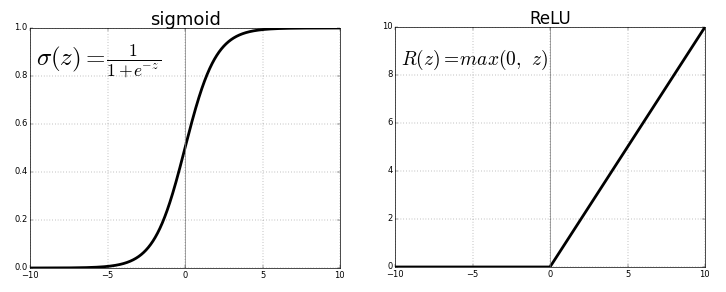
Создадим три скрытых слоя и один выходной слой нашей нейронной сети:
Python
| 1 2 3 4 | model.add(Dense(15,activation='relu')) model.add(Dense(8,activation='relu')) model.add(Dense(10,activation='relu')) model.add(Dense(1,activation='sigmoid')) |
Первые числа, передаваемые Dense, это количества нейронов, экспериментально оптимизированные в результате вариации структуры нейронной сети. Вы можете изменять количество скрытых слоев и содержащихся в них нейронов, чтобы добиться лучшего качества предсказательности модели.
Перед тем, как начать тренировать модель, ее нужно скомпилировать при помощи метода compile():
Python
| 1 | model.compile(loss="binary_crossentropy",optimizer="adam",metrics=['accuracy']) |
Методу передается три параметра:
- loss – функция потерь – объект, который модель стремиться минимизировать;
- optimizer – оптимизатор, мы используем встроенный метод стохастической оптимизации adam, описанный в публикации Дедерика Кингма и Джимми Ба;
- metrics – список метрик оптимизации, для задач классификации используется метрику ‘accuracy’.
Для обучения нейронной сети применяем метод fit():
Python
| 1 | model.fit(X,Y,epochs=1000,batch_size=10) |
Параметр epochs – «эпохи» – количество проходов нейронной сети по всем записям датасета (выбирается исходя из того, насколько быстро модель с каждым новым проходом приближается к желаемой предсказательной точности), batch_size – количество объектов выборки, берущихся за один шаг. В процессе обучения API будет выводить соответствующие строчки с величинам функции потерь и метрики для каждой из эпох.
Оценим результат обучения нейронной сети. Метод evaluate() возвращает значения функции потерь и метрики для обученной модели:
Python
| 1 2 | scores=model.evaluate(X,Y) print(" %s: %.2f%%"%(model.metrics_names[1],scores[1]*100)) |
Последняя строчка в форматированном виде выводит точность прогноза по нашей модели для заданной метрики accuracy:
| 1 | acc:87.89% |
Наконец, полный код нейронной сети с комментариями приведен в листинге ниже:
Python
| 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 | fromkeras.models importSequential fromkeras.layers importDense importnumpy # задаем для воспроизводимости результатов numpy.random.seed(2) # загружаем датасет, соответствующий последним пяти годам до определение диагноза dataset=numpy.loadtxt("prima-indians-diabetes.csv",delimiter=",") # разбиваем датасет на матрицу параметров (X) и вектор целевой переменной (Y) X,Y=dataset[:,0:8],dataset[:,8] # создаем модели, добавляем слои один за другим model=Sequential() model.add(Dense(12,input_dim=8,activation='relu'))# входной слой требует задать input_dim model.add(Dense(15,activation='relu')) model.add(Dense(8,activation='relu')) model.add(Dense(10,activation='relu')) model.add(Dense(1,activation='sigmoid'))# сигмоида вместо relu для определения вероятности # компилируем модель, используем градиентный спуск adam model.compile(loss="binary_crossentropy",optimizer="adam",metrics=['accuracy']) # обучаем нейронную сеть model.fit(X,Y,epochs=1000,batch_size=10) # оцениваем результат scores=model.evaluate(X,Y) print(" %s: %.2f%%"%(model.metrics_names[1],scores[1]*100)) |
Источник: proglib.io