Крупные подробности микроскопического мира: Нобелевская премия по химии 2017
МЕНЮ
Искусственный интеллект
Поиск
Регистрация на сайте
Помощь проекту
ТЕМЫ
Новости ИИ
Голосовой помощник
Городские сумасшедшие
ИИ в медицине
ИИ проекты
Искусственные нейросети
Слежка за людьми
Угроза ИИ
Компьютерные науки
Машинное обуч. (Ошибки)
Машинное обучение
Машинный перевод
Реализация ИИ
Реализация нейросетей
Создание беспилотных авто
Трезво про ИИ
Философия ИИ
Генетические алгоритмы
Капсульные нейросети
Основы нейронных сетей
Распознавание лиц
Распознавание образов
Распознавание речи
Техническое зрение
Чат-боты
Авторизация
2017-10-07 09:00

Люди, регулярно читающие статьи в топовых научных журналах, давно уже привыкли к многочисленным молекулярным изображениям. Но микроскопы не позволяют увидеть отдельные молекулы [1]. Правда, существует микроскопия сверхразрешения, за которую в 2014 году дали Нобелевскую премию по химии [2], но и она не позволяет «разглядеть» отдельные атомы. Изучать строение биологических молекул с такой подробностью — удел структурной биологии [3], лидирующими методиками которой до недавнего времени считались рентгеноструктурный анализ (РСА) и спектроскопия ядерного магнитного резонанса (ЯМР). Интересным методом также является атомно-силовая микроскопия [4], но герой нашего сегодняшнего рассказа другой: это криоэлектронная микроскопия, за разработку которой вручена Нобелевская премия по химии уже этого, 2017 года [5].
Визуализация сложных структур, прежде считавшихся «невидимыми» ввиду их малых размеров, позволила совершить множество фундаментальных прорывов. Один из них — прогресс в борьбе с вирусом Зика [6], [7] (рис. 1), недавно вызвавшим пандемию одноименной болезни. Благодаря криоэлектронной микроскопии, в последние пять лет все более прочно входящей в «большую тройку» методов структурной биологии, удалось получить трехмерную модель этого коварного агента. Что, в свою очередь, положило начало поиску потенциальных мишеней для лекарственных веществ, способных справиться с болезнью.
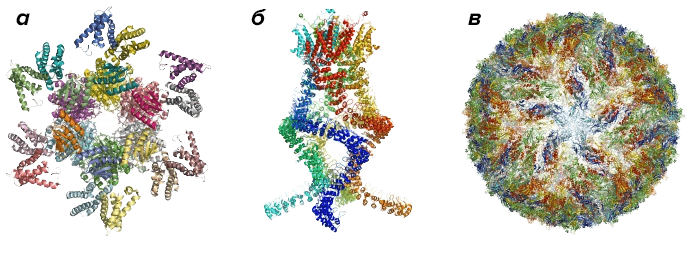
Рисунок 1. Примеры некоторых белковых комплексов. а — Белковый комплекс, регулирующий циркадный ритм. б — Комплекс звукового сенсора уха, считывающий изменения давления и позволяющий нам слышать. в — Модель вируса Зика.
Краткий экскурс в историю микроскопии до 1975 года
Всю первую половину 20 века три самых известных биологических структуры — ДНК, РНК и белок — оставались белым пятном на карте биохимического мира. Было известно, что они есть в организме и играют важную роль в жизни клеток, но каково их строение — никто не имел ни малейшего понятия. Лишь в начале 1950-х годов знаменитая группа ученых из Кембриджа, включавшая Френсиса Крика, Джеймса Уотсона, Мориса Уилкинса и Розалинд Франклин, впервые попробовала облучать ДНК рентгеновскими лучами, что привело к открытию прославленной двойной спирали.
К кристаллографам относился поначалу и Ричард Хендерсон, получивший за свои ранние исследования пусть не Нобелевскую премию, но докторскую степень. Метод рентгеноструктурного анализа заключается в дифракции рентгеновских лучей на кристаллической решетке, что помогает идентифицировать структуру молекулы. Но в то время он был еще далек от совершенства, хотя сегодня это один из основных способов изучения структуры вещества [8]. Шагнув на 30 лет вперед, мы узнаем, что наука получила еще один способ расшифровывать строение биомолекул: в 1980-х годах для изучения структуры и динамики белков в растворе начали применять метод ядерного магнитного резонанса (и применяют до сих пор [9]).
Благодаря этим двум методам удалось накопить внушительные объемы информации по строению биологических молекул: на сегодня в базе PDB находится более 100 тыс. структур. Но, как это часто бывает, и у того, и у другого метода есть свои недостатки. Так, с помощью ЯМР можно визуализировать только белки, находящиеся в растворе, и размер их должен быть небольшим. А минус рентгеновской кристаллографии считывается в названии метода: он работает на стабильных структурах типа кристаллов, но не на динамических «живых» молекулах. Изображения, полученные с помощью этого метода, напоминают снимки первых в истории фотокамер: черно-белые и застывшие, не несущие в себе информации о подвижной структуре белка. Эта проблема заставила Ричарда Хендерсона бросить рентгеновскую кристаллографию в 1970-х, что и стало отправной точкой на его пути к Нобелевской премии 2017 года.
Шаг первый: бактериородопсин под прицелом электронного пучка
Хендерсона с самого начала интересовали мембранные белки. Почему же их визуализация оказалась неподвластна рентгеновскому методу на тот момент? Неудачи возникали при попытках кристаллизовать белок, тем самым нарушая его естественное состояние в липидной мембране клетки. Мембранные белки крайне трудно извлекать из мембраны, не нарушая их нативного состояния [10]: очень часто они просто «слипаются» в единую массу, неподвластную дальнейшему изучению. Впрочем, сейчас и в кристаллизации мембранных белков есть большой прогресс: мембрану просто научились делать частью кристалла [11].
После ряда неудачных попыток Ричард Хендерсон обратился к единственному, казалось, реальному варианту: электронной микроскопии. В чем принципиальное отличие электронного микроскопа от оптического? В просвечивающей электронной микроскопии (так называется эта техника) вместо пучка света к образцу посылается луч электронов. Длина волны электронов намного меньше, чем длина волны света, поэтому с помощью электронного микроскопа можно визуализировать даже очень маленькие структуры — вплоть до уровня отдельных атомов.
В теории электронный микроскоп идеально подходил для исследований Ричарда Хендерсона — ведь он позволял получить изображения мембранных белков на атомном уровне. Но на практике эта идея казалась нереальной. Со времен изобретения электронного микроскопа считалось, что с помощью этого метода можно изучать исключительно неживую материю. Виной тому электронный пучок: он позволяет получать картинки с высоким разрешением, но фактически «сжигает» на своем пути живые структуры. Если же снизить его интенсивность, то изображение теряет контраст и получается нечетким.
Дополнительной преградой на пути визуализации биомолекул под электронным микроскопом является необходимость создания вакуума. При выкачивании воздуха из биологического образца испаряется и вода, обволакивающая живые структуры, из-за чего они теряют свою естественную форму. Таким образом, все обстоятельства выступали против Хендерсона. Однако его идею спас особый белок, обладающий исключительной стабильностью в мембране — бактериородопсин.
Бактериородопсин — светочувствительный белок, встроенный в мембрану некоторых архей. В какой-то мере его можно считать прототипом зрительного родопсина — фоторецептора нашей сетчатки [12]. Поглощая фотон, этот белок меняет конформацию, прокачивая внутрь цитоплазмы протон. Благодаря этому энергия солнечного света преобразуется в энергию разности электрических потенциалов на мембране, а затем запасается в виде энергии химических связей АТФ. Получается, что археи, подобно растениям, осуществляют фотосинтез.
Именно бактериородопсин был выбран в качестве «подопытного» объекта для исследования под электронным микроскопом. Но, в отличие от предшествующих попыток визуализации, Ричард Хендерсон с коллегами поместил под электронный пучок не экстракт белковой массы, а целый участок бактериальной мембраны. Белки, встроенные в привычную липидную среду, сохранили свою структуру. Кроме прочего, молекулы бактериородопсина образуют в мембране как бы «двумерный кристалл», создавая практически идеальные условия для изучения рентгеновской дифракции. Во избежание высыхания в вакууме исследователи зафиксировали мембранный материал в растворе глюкозы.
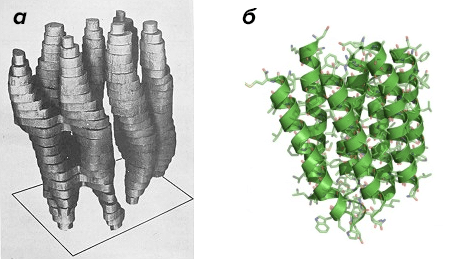
Конечно, «смертоносный» электронный луч все еще составлял проблему для исследования белка, даже под защитой мембранной упаковки. Уйти от этой помехи удалось, ослабив интенсивность пучка электронов, пропускаемых через образец. Правда, контраст изображения тоже был слабее, и отдельные молекулы были неразличимы наблюдателем. Но исследователи проанализировали дифракцию электронных лучей на белке, использовав те же принципы математического расчета, что и при рентгеноструктурном. Ученые поместили мембрану со встроенным в нее бактериородопсином под электронный микроскоп, проводя съемку с разных ракурсов. Так в 1975 году впервые удалось создать примерную трехмерную модель бактериородопсина. Этот результат стал лучшим из когда-либо созданных с помощью электронного микроскопа, но все же недостаточно впечатлил Хендерсона. Он был уверен, что электронная микроскопия способна давать гораздо большее разрешение, сравнимое с рентгеноструктурным.
В следующие несколько лет методы электронной микроскопии удалось значительно улучшить. Впервые появилась криотехнология, позволяющая исследовать образцы, охлажденные в жидком азоте, что защищало их от повреждений пучком электронов. Наконец, в 1990 году, спустя 15 лет после первой визуализации бактериородопсина, Ричарду Хендерсону удалось достигнуть своей цели: он презентовал структурную модель белка при атомном разрешении (рис. 2).
Благодаря работе Ричарда Хендерсона удалось показать, что криоэлектронная микроскопия позволяет получать изображения структуры белковых комплексов в высоком разрешении. Но как быть с белками, которые не столь идеально упорядочены, как бактериородопсин? В том же 1975 году американский ученый Йоахим Франк начал работать в этом направлении.
Шаг второй: создание одной 3D-картинки из множества 2D-изображений
Стратегия Франка основана на компьютерном анализе зашумленного изображения с распознаванием отдельных молекул белка, хаотично расположенных в мембране. Ученый разработал математический метод, благодаря которому компьютер идентифицировал на изображении молекулы в различных ракурсах и объединял их единую базу [15]. Следующий шаг — построение одной трехмерной картинки на основе множества двумерных (рис. 3). Объектом, выбранным Франком для визуализации, стала рибосома — молекулярная «машина», собирающая в клетке белки.
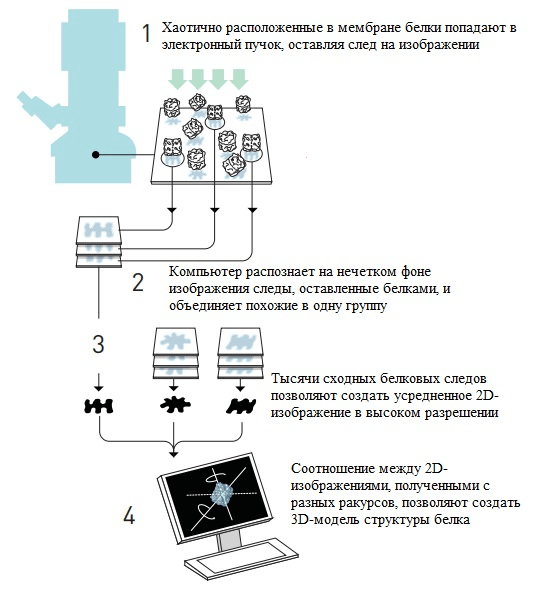
Рисунок 3. Сборка одной трехмерной картинки из множества двухмерных по методу Франка.
[5], рисунок адаптирован
Шаг третий: «остекленение» биоструктур в жидком азоте
Пока Ричард Хендерсон получал первые «фотографии» бактериородопсина, а Йоахим Франк разрабатывал математическую модель 3D-реконструкции белков из этих «фоток», другой ученый — Жак Дюбоше из Европейской молекулярно-биологической лаборатории в Гейдельберге — решал проблему «обезвоживания» биологических образцов в вакууме.
Ранее Хендерсон предложил технику фиксации образцов белков в мембране раствором глюкозы, однако такой способ не работал с молекулами, растворимыми в воде. Жак Дюбоше предложил изящное решение: предельно быстрое охлаждение воды, позволяющее ей перейти в аморфное твердое состояние, минуя стадию образования кристаллов льда — это называют витрификацией [16]. В «остекленевшей» капле воды пучок электронов рассеивается равномерно с созданием однородного фона, в котором находятся биологические молекулы. Первые опыты по криоэлектронной микроскопии были проведены с различными вирусами. Образцы помещали в воду, а затем этот раствор равномерно распределяли по металлической сетке с мелкими ячейками. Такую конструкцию замораживали в этане, охлажденном в жидком азоте, после чего образец был готов для визуализации (рис. 4). Наконец, в 1984 году Жак Дюбоше впервые опубликовал изображения вирусов разной формы, хорошо контрастирующих на прозрачном фоне остекленевшей воды (рис. 5) [17].
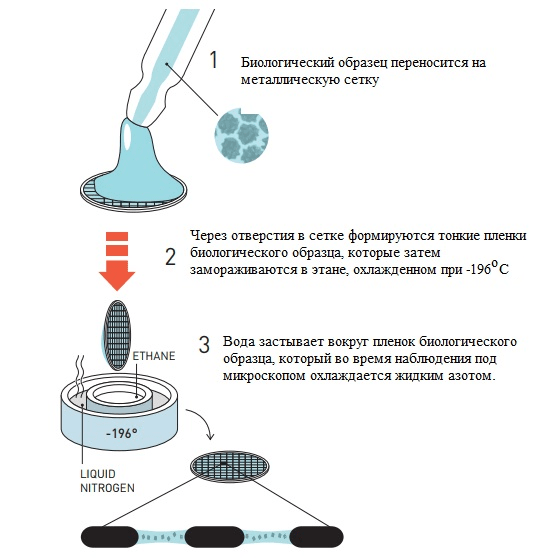
Рисунок 4. Метод заморозки биологических образцов по Дюбоше.
[5], рисунок адаптирован
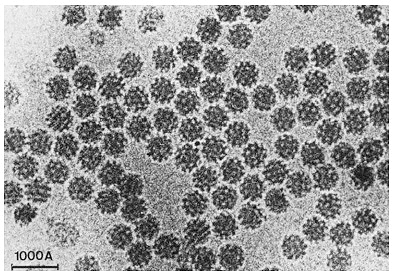
Рисунок 5. Первые микрофотографии вирусов, полученные с использованием метода Дюбоше.
В 1991 году Йоахим Франк «заморозил» рибосомы по методу Дюбоше, получив на выходе изображение их трехмерной структуры. И, несмотря на то, что картинка была сделана в небывалом для электронной микроскопии разрешении, исследователям удалось показать лишь очертания рибосомы. Странные каплевидные структуры все еще не выдерживали сравнения с атомным разрешением рентгеновской кристаллографии. Криоэлектронная микроскопия позволяла визуализировать лишь неверные контуры электронной плотности, напоминающие пузыри, из-за чего этот метод в шутку называли «блобология» (blobology) [18]. Но прогресс идет вперед, и после 2010 года получил распространение новый тип электронного детектора — Direct Electron Detector, позволяющий получать гораздо более детальное изображение биологических структур [19].
Сегодня криоэлектронная микроскопия позволяет «ловить» биологические структуры «в динамике» на разных этапах. Совмещая полученные снимки, исследователям удается создавать целые фильмы, демонстрирующие перемещения и взаимодействия белков с другими молекулами. За последние пять лет чуть ли не каждая вторая молекулярная структура, опубликованная в журналах уровня Science и Nature, криомикроскопическая: это и новые состояния рибосомы [20], и АТФазы [21], и различные рецепторы [22], [23] и филаменты загадочного тау-пептида [24], и инфламмосома [25], и термочувствительный ионный канал TRPV1 [26], и многое другое. И это только начало: ученым еще предстоит определить точную структуру и механизм работы множества белков и других сложных биологических структур.
Литература
- Методы в картинках: микроскопия;
- По ту сторону дифракционного барьера: Нобелевская премия по химии 2014;
- Методы в картинках: структурная биология;
- Атомно-силовая микроскопия: увидеть, прикоснувшись;
- Fernholm A. (2017). They captured life in atomic detail. Nobelprize.org;
- Вирус Зика — глобальная угроза?;
- Вирус Зика: мнимая паника или реальная угроза?;
- Фемтосекундные рентгеновские лазеры — кристаллография будущего;
- Сверхпроводящие магниты и рецепторы биомембран: Лаборатория биомолекулярной ЯМР-спектроскопии ИБХ РАН;
- Липидный фундамент жизни;
- Лаборатория перспективных исследований мембранных белков: от гена к ангстрему;
- Зрительный родопсин — рецептор, реагирующий на свет;
- R. Henderson, P. N. T. Unwin. (1975). Three-dimensional model of purple membrane obtained by electron microscopy. Nature. 257, 28-32;
- R. Henderson, J.M. Baldwin, T.A. Ceska, F. Zemlin, E. Beckmann, K.H. Downing. (1990). Model for the structure of bacteriorhodopsin based on high-resolution electron cryo-microscopy. Journal of Molecular Biology. 213, 899-929;
- Joachim Frank. (1975). Averaging of low exposure electron micrographs of non-periodic objects. Ultramicroscopy. 1, 159-162;
- Витрификация — контролируемая пауза развития в стеклоподобном состоянии;
- Marc Adrian, Jacques Dubochet, Jean Lepault, Alasdair W. McDowall. (1984). Cryo-electron microscopy of viruses. Nature. 308, 32-36;
- Канал эукариотического шаперонина открывается подобно диафрагме фотоаппарата;
- Anna-Clare Milazzo, Philippe Leblanc, Fred Duttweiler, Liang Jin, James C. Bouwer, et. al.. (2005). Active pixel sensor array as a detector for electron microscopy. Ultramicroscopy. 104, 152-159;
- Niels Fischer, Piotr Neumann, Andrey L. Konevega, Lars V. Bock, Ralf Ficner, et. al.. (2015). Structure of the E. coli ribosome–EF-Tu complex at <3 ? resolution by Cs-corrected cryo-EM. Nature. 520, 567-570;
- Matteo Allegretti, Niklas Klusch, Deryck J. Mills, Janet Vonck, Werner K?hlbrandt, Karen M. Davies. (2015). Horizontal membrane-intrinsic ?-helices in the stator a-subunit of an F-type ATP synthase. Nature. 521, 237-240;
- Tian Hua, Kiran Vemuri, Spyros P. Nikas, Robert B. Laprairie, Yiran Wu, et. al.. (2017). Crystal structures of agonist-bound human cannabinoid receptor CB1. Nature;
- B. Herguedas, J. Garcia-Nafria, O. Cais, R. Fernandez-Leiro, J. Krieger, et. al.. (2016). Structure and organization of heteromeric AMPA-type glutamate receptors. Science. 352, aad3873-aad3873;
- Anthony W. P. Fitzpatrick, Benjamin Falcon, Shaoda He, Alexey G. Murzin, Garib Murshudov, et. al.. (2017). Cryo-EM structures of tau filaments from Alzheimer’s disease. Nature. 547, 185-190;
- L. Zhang, S. Chen, J. Ruan, J. Wu, A. B. Tong, et. al.. (2015). Cryo-EM structure of the activated NAIP2-NLRC4 inflammasome reveals nucleated polymerization. Science. 350, 404-409;
- Yuan Gao, Erhu Cao, David Julius, Yifan Cheng. (2016). TRPV1 structures in nanodiscs reveal mechanisms of ligand and lipid action. Nature. 534, 347-351.
Источник: biomolecula.ru